Modèle épidémiologique (SIRD)¶
Etrangement, il n’était pas possible d’accéder aux données de l’épidémie le jour du TD car le site data.gouv.fr était inacessible suite à une panne chez OVH.
Comme les vraies données sont inaccessibles, on s’intéresse à des données simulées.
Simulation¶
[3]:
import numpy
def iteration(S, I, R, D, beta, mu, nu, N):
dR = I * mu
dD = I * nu
dS = -S * I * beta / N
dI = -dS - dR - dD
return S + dS, I + dI, R + dR, D + dD
iteration(10, 1, 0, 0, 0.1, 0.1, 0.1, 11)
[3]:
(9.909090909090908, 0.8909090909090909, 0.1, 0.1)
[4]:
def simulation(n_iter, S, I, R, D, beta, mu, nu):
sim = numpy.zeros((n_iter + 1, 4))
sim[0, :] = numpy.array([S, I, R, D]).ravel()
N = S + I + R + D
for i in range(n_iter):
S, I, R, D = iteration(S, I, R, D, beta, mu, nu, N)
sim[i + 1, :] = numpy.array([S, I, R, D]).ravel()
return sim
simulation(2, 10, 1, 0, 0, 0.1, 0.1, 0.1)
[4]:
array([[10. , 1. , 0. , 0. ],
[ 9.90909091, 0.89090909, 0.1 , 0.1 ],
[ 9.82883546, 0.79298272, 0.18909091, 0.18909091]])
Visualisation¶
[5]:
%matplotlib inline
[7]:
import matplotlib.pyplot as plt
import pandas
fig, ax = plt.subplots(1, 1, figsize=(10, 2))
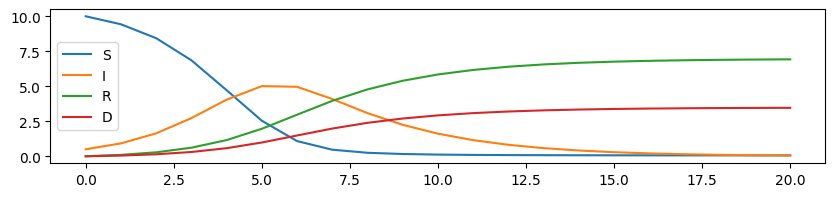
sim = simulation(20, 10, 0.5, 0, 0, 1.2, 0.2, 0.1)
df = pandas.DataFrame(sim, columns=["S", "I", "R", "D"])
df.plot(ax=ax);
Estimation des paramètres¶
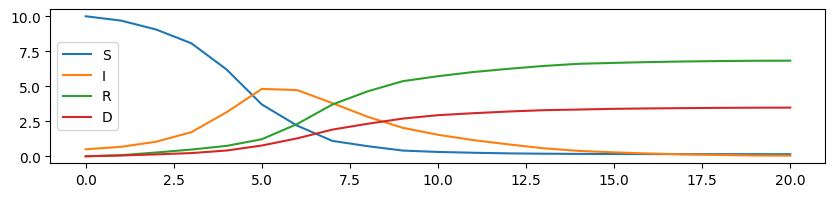
Une fois qu’on a des données, on peut essayer de retrouver la paramètres de la simulation. Mais auparavant, on simule des données un peu plus bruitées en changeant les paramètres du modèle tous les jours.
[8]:
def simulation_bruitee(n_iter, S, I, R, D, beta, mu, nu):
sim = numpy.zeros((n_iter + 1, 4))
sim[0, :] = numpy.array([S, I, R, D]).ravel()
N = S + I + R + D
for i in range(n_iter):
b = numpy.random.randn(1) * beta / 5 + beta
m = numpy.random.randn(1) * mu / 5 + mu
n = numpy.random.randn(1) * nu / 5 + nu
S, I, R, D = iteration(S, I, R, D, b, m, n, N)
sim[i + 1, :] = numpy.array([S, I, R, D]).ravel()
return sim
simulation_bruitee(2, 10, 1, 0, 0, 0.1, 0.1, 0.1)
[8]:
array([[10. , 1. , 0. , 0. ],
[ 9.90629495, 0.90818313, 0.0769856 , 0.10853633],
[ 9.84322669, 0.77113049, 0.17022418, 0.21541865]])
[9]:
fig, ax = plt.subplots(1, 1, figsize=(10, 2))
sim_bruit = simulation_bruitee(20, 10, 0.5, 0, 0, 1.2, 0.2, 0.1)
df = pandas.DataFrame(sim_bruit, columns=["S", "I", "R", "D"])
df.plot(ax=ax);
Première idée¶
On calcule les paramètres chaque jour à partir de la définition du modèle.
[10]:
def estimation_coefficient(sim):
N = sim.sum(axis=1)
diff = sim[1:] - sim[:-1]
mu = diff[:, 2] / sim[:-1, 1]
nu = diff[:, 3] / sim[:-1, 1]
beta = -diff[:, 0] / (sim[:-1, 1] * sim[:-1, 0]) * N[:-1]
return dict(beta=beta, mu=mu, nu=nu)
df = pandas.DataFrame(estimation_coefficient(sim_bruit))
df.head(n=1)
[10]:
| beta | mu | nu | |
|---|---|---|---|
| 0 | 0.645167 | 0.153483 | 0.113224 |
[11]:
df.plot(figsize=(10, 2));
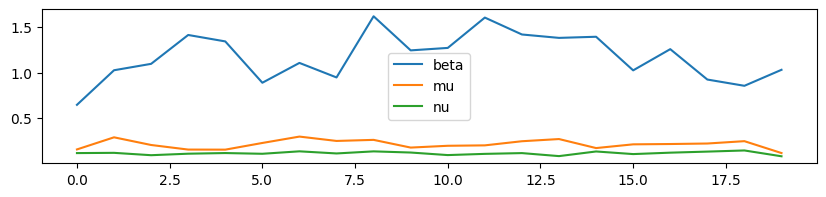
On pourrait imaginer que les paramètres cherchés soient égaux à la moyenne des valeurs obtenues pour chaque jour. Encore faudrait-il le prouver ou trouver un contre exemple.
Seconde idée : problèmes d’optimisation¶
On cherche à transformer le problème d’estimation en un problème d’optimisation de telle sorte que les paramètres en sont la solution. L’idée de simuler en fonction d’un jeu de paramètres puis de calculer une distance entre cette simulation et les données observées.
Le problème de cette solution est qu’il faut essayer plein de valeur, au hasard pour faire simple. Donc on se sert de la fonction précédente comme point de départ et on tire des valeurs tout autour.
[12]:
def distance_sim(obs, sim):
return ((obs - sim) ** 2).sum() / obs.size
distance_sim(sim, sim_bruit)
[12]:
0.1379327480825296
[13]:
def optimisation(obs):
est = estimation_coefficient(obs)
b0 = est["beta"].mean()
m0 = est["mu"].mean()
n0 = est["nu"].mean()
n_iter = obs.shape[0] - 1
S, I, R, D = obs[0]
beta, mu, nu = b0, m0, n0
distance = distance_sim(obs, simulation(n_iter, S, I, R, D, b0, m0, n0))
distance0 = distance
for i in range(0, 100):
b = numpy.random.randn(1) * b0 / 5 + b0
m = numpy.random.randn(1) * m0 / 5 + m0
n = numpy.random.randn(1) * n0 / 5 + n0
sim = simulation(n_iter, S, I, R, D, b, m, n)
d = distance_sim(obs, sim)
if distance is None or d < distance:
distance = d
beta, mu, nu = b, m, n
return dict(
b0=b0,
mu0=m0,
nu0=n0,
beta=beta,
mu=mu,
nu=nu,
distance=distance,
distance0=distance0,
)
optimisation(sim_bruit)
[13]:
{'b0': 1.175492030437819,
'mu0': 0.21093122422461147,
'nu0': 0.11141742761811038,
'beta': array([1.12240477]),
'mu': array([0.1995322]),
'nu': array([0.09668045]),
'distance': 0.06764482535407212,
'distance0': 0.0880922396685696}
distance0 la distance entre la simulation obtenue avec les paramètes de la première méthode, distance celle de la seconde et elle est plus petite. Cela répond à la question qu’on se posait ci-dessus.
[ ]: