DataFrame et Graphes¶
Les Dataframe se sont imposés pour manipuler les données. Avec cette façon de représenter les données, associée à des méthodes couramment utilisées, ce qu’on faisait en une ou deux boucles se fait maintenant en une seule fonction. Le module pandas est très utilisé, il existe de nombreux tutoriels, ou page de recettes pour les usages les plus fréquents : cookbook.
[1]:
%matplotlib inline
# Cette première instruction indique à Jupyter d'insérer les graphiques
# dans le notebook plutôt que dans une fenêtre externe.
import matplotlib.pyplot as plt
La première instruction %matplotlib est spécifique aux notebooks. Sans cela, les graphiques ne sont pas insérées dans la page elle-même mais cela ne s’applique qu’aux notebooks et provoquera une erreur sur Spyder par exemple.
DataFrame¶
Pour faire court, c’est l’équivalent d’une feuille Excel ou d’une table SQL.
Taille de DataFrame
Les DataFrame en Python sont assez rapides lorsqu’il y a moins de 10 millions d’observations et que le fichier texte qui décrit les données n’est pas plus gros que 10 Mo. Au delà, il faut soit être patient, soit être astucieux, utiliser sqllite et passer au SQL ou parcourir les données en streaming.
Valeurs manquantes
Lorsqu’on récupère des données, il peut arriver qu’une valeur soit manquante (Working with missing data).
[ ]:
from IPython.display import Image
Image("td2df.png")
Pour manipuler les dataframe, on utilise le module pandas. Il est prévu pour manipuler les données d’une table par bloc (une sous-table). Tant qu’on manipule des blocs, le module est rapide.
Series¶
Une Series est un objet uni-dimensionnel similaire à un tableau, une liste ou une colonne d’une table. Chaque valeur est associée à un index qui est par défaut les entiers de 0 à (avec
la longueur de la Series).
[3]:
import pandas
from pandas import Series
import numpy
s = Series([42, "Hello World!", 3.14, -5, None, numpy.nan])
s.head()
[3]:
0 42
1 Hello World!
2 3.14
3 -5
4 None
dtype: object
On peut aussi préciser les indices lors de la création, ou construire la Series à partir d’un dictionnaire si on fournit un index avec un dictionnaire, les index qui ne sont pas des clés du dictionnaire seront des valeurs manquantes.
[4]:
s2 = Series(
[42, "Hello World!", 3.14, -5, None, numpy.nan],
index=["int", "string", "pi", "neg", "missing1", "missing2"],
)
city2cp_dict = {"Paris14": 75014, "Paris18": 75018, "Malakoff": 92240, "Nice": 6300}
cities = Series(city2cp_dict)
cities
[4]:
Paris14 75014
Paris18 75018
Malakoff 92240
Nice 6300
dtype: int64
Quelques liens pour comprendre le code suivant : Series.isnull, Series.notnull.
[5]:
cities_list = ["Paris12"] + list(city2cp_dict.keys()) + ["Vanves"]
cities2 = Series(city2cp_dict, index=cities_list)
pandas.isnull(cities2) # same as cities2.isnull()
pandas.notnull(cities2)
[5]:
Paris12 False
Paris14 True
Paris18 True
Malakoff True
Nice True
Vanves False
dtype: bool
On peut se servir de l’index pour sélectionner une ou plusieurs valeurs de la Series, éventuellement pour en changer la valeur. On peut aussi appliquer des opérations mathématiques, filtrer avec un booléen, ou encore tester la présence d’un élement.
[ ]:
cities2["Nice"] # renvoie un scalaire
cities2[["Malakoff", "Paris14"]] # renvoie une Series
cities2["Paris12"] = 75012
dep = cities2 // 1000 # // pour une division entière
dep
Paris12 75.0
Paris14 75.0
Paris18 75.0
Malakoff 92.0
Nice 6.0
Vanves NaN
dtype: float64
pandas aligne automatiquement les données en utilisant l’index des Series lorsqu’on fait une opération sur des series.
[ ]:
cities2[dep == 75]
print("Paris14", "Paris14" in cities2)
print("Paris13", "Paris13" in cities2)
Paris14 True
Paris13 False
[ ]:
# print(cities)
# print(cities2)
cities + cities2
Malakoff 184480.0
Nice 12600.0
Paris12 NaN
Paris14 150028.0
Paris18 150036.0
Vanves NaN
dtype: float64
pandas garde les lignes communes aux deux tables et additionnent les colonnes portant le même nom. On peut nommer la Series, ses index et même assigner un nouvel index à une Series existante.
[ ]:
cities2.name = "Code Postal"
cities2.index.name = "Ville"
print(cities2)
print("-------------")
s2.index = range(6)
print(s2)
Ville
Paris12 75012.0
Paris14 75014.0
Paris18 75018.0
Malakoff 92240.0
Nice 6300.0
Vanves NaN
Name: Code Postal, dtype: float64
-------------
0 42
1 Hello World!
2 3.14
3 -5
4 None
5 NaN
dtype: object
DataFrame (pandas)¶
Quelques liens : An Introduction to Pandas
Un DataFrame est un objet qui est présent dans la plupart des logiciels de traitements de données, c’est une matrice, chaque colonne est une Series et est de même type (nombre, date, texte), elle peut contenir des valeurs manquantes (nan). On peut considérer chaque colonne comme les variables d’une table (pandas.Dataframe - cette page contient toutes les méthodes de la classe).
Un Dataframe représente une table de données, i.e. une collection ordonnées de colonnes. Ces colonnes/lignes peuvent avoir des types différents (numérique, string, boolean). Cela est très similaire aux DataFrame du langage R (en apparence…), avec un traitement plus symétrique des lignes et des colonnes.
[10]:
import pandas
l = [
{"date": "2014-06-22", "prix": 220.0, "devise": "euros"},
{"date": "2014-06-23", "prix": 221.0, "devise": "euros"},
]
df = pandas.DataFrame(l)
df
[10]:
| date | prix | devise | |
|---|---|---|---|
| 0 | 2014-06-22 | 220.0 | euros |
| 1 | 2014-06-23 | 221.0 | euros |
Avec une valeur manquante :
[ ]:
l = [
{"date": "2014-06-22", "prix": 220.0, "devise": "euros"},
{"date": "2014-06-23", "devise": "euros"},
]
df = pandas.DataFrame(l)
df
| date | prix | devise | |
|---|---|---|---|
| 0 | 2014-06-22 | 220.0 | euros |
| 1 | 2014-06-23 | NaN | euros |
NaN est une convention pour une valeur manquante. On extrait la variable prix :
[ ]:
df.prix
0 220.0
1 NaN
Name: prix, dtype: float64
Ou :
[ ]:
df["prix"]
0 220.0
1 NaN
Name: prix, dtype: float64
Pour extraire plusieurs colonnes :
[14]:
df[["date", "prix"]]
[14]:
| date | prix | |
|---|---|---|
| 0 | 2014-06-22 | 220.0 |
| 1 | 2014-06-23 | NaN |
Pour prendre la transposée (voir aussi DataFrame.transpose) :
[ ]:
df.T
| 0 | 1 | |
|---|---|---|
| date | 2014-06-22 | 2014-06-23 |
| prix | 220.0 | NaN |
| devise | euros | euros |
Lecture et écriture de DataFrame¶
Aujourd’hui, on n’a plus besoin de réécrire soi-même une fonction de lecture ou d’écriture de données présentées sous forme de tables. Il existe des fonctions plus génériques qui gère un grand nombre de cas. Cette section présente brièvement les fonctions qui permettent de lire/écrire un DataFrame aux formats texte/Excel. On reprend l’exemple de
section précédente. L’instruction encoding=utf-8 n’est pas obligatoire mais conseillée lorsque les données contiennent des accents (voir read_csv).
[16]:
import pandas
l = [
{"date": "2014-06-22", "prix": 220.0, "devise": "euros"},
{"date": "2014-06-23", "prix": 221.0, "devise": "euros"},
]
df = pandas.DataFrame(l)
# écriture au format texte
df.to_csv("exemple.txt", sep="\t", encoding="utf-8", index=False)
# on regarde ce qui a été enregistré
with open("exemple.txt", "r", encoding="utf-8") as f:
text = f.read()
print(text)
# on enregistre au format Excel
df.to_excel("exemple.xlsx", index=False)
# special jupyter - notebook
%system "exemple.xlsx"
date prix devise
2014-06-22 220.0 euros
2014-06-23 221.0 euros
[16]:
['/bin/bash: line 1: exemple.xlsx: command not found']
On peut récupérer des données directement depuis Internet ou une chaîne de caractères et afficher le début (head) ou la fin (tail).
[17]:
import pandas
df = df_marathon = pandas.read_csv(
"https://github.com/sdpython/teachpyx/raw/main/_data/marathon.txt",
sep="\t",
names=["ville", "annee", "temps", "secondes"],
)
df.head()
[17]:
| ville | annee | temps | secondes | |
|---|---|---|---|---|
| 0 | PARIS | 2011 | 02:06:29 | 7589 |
| 1 | PARIS | 2010 | 02:06:41 | 7601 |
| 2 | PARIS | 2009 | 02:05:47 | 7547 |
| 3 | PARIS | 2008 | 02:06:40 | 7600 |
| 4 | PARIS | 2007 | 02:07:17 | 7637 |
La fonction describe permet d’en savoir un peu plus sur les colonnes numériques de cette table.
[18]:
df.describe()
[18]:
| annee | secondes | |
|---|---|---|
| count | 359.000000 | 359.000000 |
| mean | 1989.754875 | 7933.660167 |
| std | 14.028545 | 385.289830 |
| min | 1947.000000 | 7382.000000 |
| 25% | 1981.000000 | 7698.000000 |
| 50% | 1991.000000 | 7820.000000 |
| 75% | 2001.000000 | 8046.500000 |
| max | 2011.000000 | 10028.000000 |
DataFrame et Index¶
On désigne généralement une colonne ou variable par son nom. Les lignes peuvent être désignées par un entier.
[ ]:
import pandas
l = [
{"date": "2014-06-22", "prix": 220.0, "devise": "euros"},
{"date": "2014-06-23", "prix": 221.0, "devise": "euros"},
]
df = pandas.DataFrame(l)
df
| date | prix | devise | |
|---|---|---|---|
| 0 | 2014-06-22 | 220.0 | euros |
| 1 | 2014-06-23 | 221.0 | euros |
On extrait une ligne avec (iloc).
[ ]:
df.iloc[1]
date 2014-06-23
prix 221.0
devise euros
Name: 1, dtype: object
On extrait une colonne avec [loc]( ou iloc.
[ ]:
df.loc[1]
date 2014-06-23
prix 221.0
devise euros
Name: 1, dtype: object
On extrait une valeur en indiquant sa position dans la table avec des entiers :
[ ]:
df.iloc[1, 2]
'euros'
Avec loc, il faut préciser le nombre de la colonne.
[ ]:
df.columns
Index(['date', 'prix', 'devise'], dtype='str')
[ ]:
df.loc[1, "prix"]
np.float64(221.0)
Mais il est possible d’utiliser une colonne ou plusieurs colonnes comme index (set_index) :
[25]:
dfi = df.set_index("date")
dfi
[25]:
| prix | devise | |
|---|---|---|
| date | ||
| 2014-06-22 | 220.0 | euros |
| 2014-06-23 | 221.0 | euros |
On peut maintenant désigner une ligne par une date avec loc (mais pas iloc car iloc n’accepte que des entiers qui se réfère aux index de chaque dimension).
[ ]:
dfi.loc["2014-06-23"]
prix 221.0
devise euros
Name: 2014-06-23, dtype: object
Il est possible d’utiliser plusieurs colonnes comme index :
[ ]:
df = pandas.DataFrame(
[
{"prénom": "xavier", "nom": "dupré", "arrondissement": 18},
{"prénom": "clémence", "nom": "dupré", "arrondissement": 15},
]
)
dfi = df.set_index(["nom", "prénom"])
dfi.loc["dupré", "xavier"]
arrondissement 18
Name: (dupré, xavier), dtype: int64
Si on veut changer l’index ou le supprimer (reset_index) :
[ ]:
dfi.reset_index(drop=False, inplace=True)
# le mot-clé drop pour garder ou non les colonnes servant d'index
# inplace signifie qu'on modifie l'instance et non qu'une copie est modifiée
# donc on peut aussi écrire dfi2 = dfi.reset_index(drop=False)
dfi.set_index(["nom", "arrondissement"], inplace=True)
dfi
| prénom | ||
|---|---|---|
| nom | arrondissement | |
| dupré | 18 | xavier |
| 15 | clémence |
Les index sont particulièrement utiles lorsqu’il s’agit de fusionner deux tables. Pour des petites tables, la plupart du temps, il est plus facile de s’en passer.
Notation avec le symbole :¶
Le symbole : désigne une plage de valeurs.
[ ]:
import pandas, urllib.request
df = df_marathon
df.head()
| ville | annee | temps | secondes | |
|---|---|---|---|---|
| 0 | PARIS | 2011 | 02:06:29 | 7589 |
| 1 | PARIS | 2010 | 02:06:41 | 7601 |
| 2 | PARIS | 2009 | 02:05:47 | 7547 |
| 3 | PARIS | 2008 | 02:06:40 | 7600 |
| 4 | PARIS | 2007 | 02:07:17 | 7637 |
On peut sélectionner un sous-ensemble de lignes :
[ ]:
df[3:6]
| ville | annee | temps | secondes | |
|---|---|---|---|---|
| 3 | PARIS | 2008 | 02:06:40 | 7600 |
| 4 | PARIS | 2007 | 02:07:17 | 7637 |
| 5 | PARIS | 2006 | 02:08:03 | 7683 |
On extrait la même plage mais avec deux colonnes seulement :
[ ]:
df.loc[3:6, ["annee", "temps"]]
| annee | temps | |
|---|---|---|
| 3 | 2008 | 02:06:40 |
| 4 | 2007 | 02:07:17 |
| 5 | 2006 | 02:08:03 |
| 6 | 2005 | 02:08:02 |
Le même code pour lequel on renomme les colonnes extraites :
[ ]:
sub = df.loc[3:6, ["annee", "temps"]]
sub.columns = ["year", "time"]
sub
| year | time | |
|---|---|---|
| 3 | 2008 | 02:06:40 |
| 4 | 2007 | 02:07:17 |
| 5 | 2006 | 02:08:03 |
| 6 | 2005 | 02:08:02 |
Exercice 1 : créer un fichier Excel¶
On souhaite récupérer les données donnees_enquete_2003_television.txt.
POIDSLOG: Pondération individuelle relativePOIDSF: Variable de pondération individuellecLT1FREQ: Nombre d’heures en moyenne passées à regarder la télévisioncLT2FREQ: Unité de temps utilisée pour compter le nombre d’heures passées à regarder la télévision, cette unité est représentée par les quatre valeurs suivantes0 : non concerné
1 : jour
2 : semaine
3 : mois
Ensuite, on veut :
Supprimer les colonnes vides
Obtenir les valeurs distinctes pour la colonne
cLT2FREQModifier la matrice pour enlever les lignes pour lesquelles l’unité de temps (cLT2FREQ) n’est pas renseignée ou égale à zéro.
Sauver le résultat au format Excel.
Vous aurez peut-être besoin des fonctions suivantes :
[ ]:
import pandas, io
# ...
Manipuler un DataFrame¶
Si la structure DataFrame s’est imposée, c’est parce qu’on effectue toujours les mêmes opérations. Chaque fonction cache une boucle ou deux dont le coût est précisé en fin de ligne :
filter : on sélectionne un sous-ensemble de lignes qui vérifie une condition
union : concaténation de deux jeux de données
sort : tri
group by : grouper des lignes qui partagent une valeur commune
join : fusionner deux jeux de données en associant les lignes qui partagent une valeur commune
pivot : utiliser des valeurs présentes dans colonne comme noms de colonnes
Les 5 premières opérations sont issues de la logique de manipulation des données avec le langage SQL (ou le logiciel SAS). La dernière correspond à un tableau croisé dynamique. Pour illustrer ces opérations, on prendre le DataFrame suivant :
[ ]:
import pandas
df = df_marathon
print(df.columns)
print("villes", set(df.ville))
print("annee", list(set(df.annee))[:10], "...")
Index(['ville', 'annee', 'temps', 'secondes'], dtype='str')
villes {'AMSTERDAM', 'FUKUOKA', 'BERLIN', 'LONDON', 'STOCKOLM', 'NEW YORK', 'PARIS', 'BOSTON', 'CHICAGO'}
annee [1947, 1948, 1949, 1950, 1951, 1952, 1953, 1954, 1955, 1956] ...
6 opérations : filtrer, union, sort, group by, join, pivot¶
filter¶
Filter consiste à sélectionner un sous-ensemble de lignes du dataframe. Pour filter sur plusieurs conditions, il faut utiliser les opérateurs logique & (et), | (ou), ~ (non) (voir Mapping Operators to Functions).
[35]:
subset = df[df.annee == 1971]
subset.head()
[35]:
| ville | annee | temps | secondes | |
|---|---|---|---|---|
| 112 | FUKUOKA | 1971 | 02:12:51 | 7971 |
| 204 | NEW YORK | 1971 | 02:22:54 | 8574 |
| 285 | BOSTON | 1971 | 02:18:45 | 8325 |
[36]:
subset = df[(df.annee == 1971) & (df.ville == "BOSTON")]
subset.head()
[36]:
| ville | annee | temps | secondes | |
|---|---|---|---|---|
| 285 | BOSTON | 1971 | 02:18:45 | 8325 |
Les dernières versions de pandas ont introduit la méthode query qui permet de réduire encore l’écriture :
[ ]:
subset = df.query('(annee == 1971) & (ville == "BOSTON")')
subset.head()
| ville | annee | temps | secondes | |
|---|---|---|---|---|
| 285 | BOSTON | 1971 | 02:18:45 | 8325 |
union¶
union = concaténation de deux DataFrame (qui n’ont pas nécessairement les mêmes colonnes). On peut concaténer les lignes ou les colonnes.
[ ]:
concat_ligne = pandas.concat((df, df))
df.shape, concat_ligne.shape
((359, 4), (718, 4))
[ ]:
concat_col = pandas.concat((df, df), axis=1)
df.shape, concat_col.shape
((359, 4), (359, 8))
sort¶
Sort = trier
[ ]:
tri = df.sort_values(["annee", "ville"], ascending=[0, 1])
tri.head()
| ville | annee | temps | secondes | |
|---|---|---|---|---|
| 35 | BERLIN | 2011 | 02:03:38 | 7418 |
| 325 | BOSTON | 2011 | 02:03:02 | 7382 |
| 202 | LONDON | 2011 | 02:04:40 | 7480 |
| 0 | PARIS | 2011 | 02:06:29 | 7589 |
| 276 | STOCKOLM | 2011 | 02:14:07 | 8047 |
group by¶
Cette opération consiste à grouper les lignes qui partagent une caractéristique commune (une valeur dans une colonne ou plusieurs valeurs dans plusieurs colonnes). On peut conserver chaque groupe, ou calculer une somme, une moyenne, prendre la ou meilleures valeurs (top per group)…
[ ]:
gr = df.groupby("annee")
gr
<pandas.api.typing.DataFrameGroupBy object at 0x78ca1676a9c0>
[ ]:
nb = df.groupby("annee").count()
nb.sort_index(ascending=False).head()
| ville | temps | secondes | |
|---|---|---|---|
| annee | |||
| 2011 | 5 | 5 | 5 |
| 2010 | 9 | 9 | 9 |
| 2009 | 9 | 9 | 9 |
| 2008 | 9 | 9 | 9 |
| 2007 | 9 | 9 | 9 |
[ ]:
nb = df[["annee", "secondes"]].groupby("annee").sum()
nb.sort_index(ascending=False).head(n=2)
| secondes | |
|---|---|
| annee | |
| 2011 | 37916 |
| 2010 | 68673 |
[ ]:
nb = df[["annee", "secondes"]].groupby("annee").mean()
nb.sort_index(ascending=False).head(n=3)
| secondes | |
|---|---|
| annee | |
| 2011 | 7583.200000 |
| 2010 | 7630.333333 |
| 2009 | 7652.555556 |
Si les nom des colonnes utilisées lors de l’opération ne sont pas mentionnés, implicitement, c’est l’index qui sera choisi. On peut aussi aggréger les informations avec une fonction personnalisée.
[ ]:
def max_entier(x):
return int(max(x))
nb = df[["annee", "secondes"]].groupby("annee").agg(max_entier).reset_index()
nb.tail(n=3)
| annee | secondes | |
|---|---|---|
| 62 | 2009 | 8134 |
| 63 | 2010 | 7968 |
| 64 | 2011 | 8047 |
Ou encore considérer des aggrégations différentes pour chaque colonne :
[ ]:
nb = (
df[["annee", "ville", "secondes"]]
.groupby("annee")
.agg({"ville": len, "secondes": max_entier})
)
nb.tail(n=3)
| ville | secondes | |
|---|---|---|
| annee | ||
| 2009 | 9 | 8134 |
| 2010 | 9 | 7968 |
| 2011 | 5 | 8047 |
On veut extraire les deux meilleurs temps par ville :
[ ]:
series = df.groupby(["ville"]).apply(lambda r: r["secondes"].nsmallest(2))
[ ]:
indices = [t[1] for t in series.index]
indices
[171,
170,
35,
38,
325,
324,
357,
347,
74,
75,
202,
200,
234,
222,
2,
0,
248,
251]
[49]:
df.loc[indices]
[49]:
| ville | annee | temps | secondes | |
|---|---|---|---|---|
| 171 | AMSTERDAM | 2010 | 02:05:44 | 7544 |
| 170 | AMSTERDAM | 2009 | 02:06:18 | 7578 |
| 35 | BERLIN | 2011 | 02:03:38 | 7418 |
| 38 | BERLIN | 2008 | 02:03:59 | 7439 |
| 325 | BOSTON | 2011 | 02:03:02 | 7382 |
| 324 | BOSTON | 2010 | 02:05:52 | 7552 |
| 357 | CHICAGO | 2009 | 02:05:41 | 7541 |
| 347 | CHICAGO | 1999 | 02:05:42 | 7542 |
| 74 | FUKUOKA | 2009 | 02:05:18 | 7518 |
| 75 | FUKUOKA | 2008 | 02:06:10 | 7570 |
| 202 | LONDON | 2011 | 02:04:40 | 7480 |
| 200 | LONDON | 2009 | 02:05:10 | 7510 |
| 234 | NEW YORK | 2001 | 02:07:43 | 7663 |
| 222 | NEW YORK | 1989 | 02:08:01 | 7681 |
| 2 | PARIS | 2009 | 02:05:47 | 7547 |
| 0 | PARIS | 2011 | 02:06:29 | 7589 |
| 248 | STOCKOLM | 1983 | 02:11:37 | 7897 |
| 251 | STOCKOLM | 1986 | 02:12:33 | 7953 |
join (merge ou fusion)¶
Fusionner deux tables consiste à apparier les lignes de la première table avec celle de la seconde si certaines colonnes de ces lignes partagent les mêmes valeurs. On distingue quatre cas :
INNER JOIN- inner : on garde tous les appariements réussisLEFT OUTER JOIN- left : on garde tous les appariements réussis et les lignes non appariées de la table de gaucheRIGHT OUTER JOIN- right : on garde tous les appariements réussis et les lignes non appariées de la table de droiteFULL OUTER JOIN- outer : on garde tous les appariements réussis et les lignes non appariées des deux tables
Exemples et documentation :
jointures SQL - illustrations avec graphiques en patates
Si les noms des colonnes utilisées lors de la fusion ne sont pas mentionnés, implicitement, c’est l’index qui sera choisi. Pour les grandes tables (> 1.000.000 lignes), il est fortement recommandé d’ajouter un index s’il n’existe pas avant de fusionner. A quoi correspondent les quatre cas suivants (LEFT ou FULL ou RIGHT ou INNER) ?
[ ]:
from IPython.display import Image
Image("patates.png")
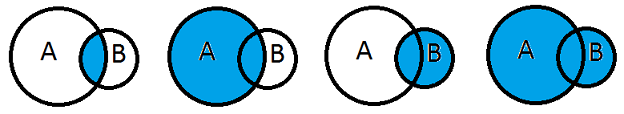
On souhaite ajouter une colonne pays aux marathons se déroulant dans les villes suivantes.
[ ]:
values = [
{"V": "BOSTON", "C": "USA"},
{"V": "NEW YORK", "C": "USA"},
{"V": "BERLIN", "C": "Germany"},
{"V": "LONDON", "C": "UK"},
{"V": "PARIS", "C": "France"},
]
pays = pandas.DataFrame(values)
pays
| V | C | |
|---|---|---|
| 0 | BOSTON | USA |
| 1 | NEW YORK | USA |
| 2 | BERLIN | Germany |
| 3 | LONDON | UK |
| 4 | PARIS | France |
[52]:
dfavecpays = df.merge(pays, left_on="ville", right_on="V")
pandas.concat([dfavecpays.head(n=2), dfavecpays.tail(n=2)])
[52]:
| ville | annee | temps | secondes | V | C | |
|---|---|---|---|---|---|---|
| 0 | PARIS | 2011 | 02:06:29 | 7589 | PARIS | France |
| 1 | PARIS | 2010 | 02:06:41 | 7601 | PARIS | France |
| 192 | BOSTON | 2010 | 02:05:52 | 7552 | BOSTON | USA |
| 193 | BOSTON | 2011 | 02:03:02 | 7382 | BOSTON | USA |
Question :
Que changerait l’ajout du paramètre ``how=”outer”`` dans ce cas ?
On cherche à joindre deux tables A,B qui ont chacune trois clés distinctes : :math:`c_1, c_2, c_3`. Il y a respectivement dans chaque table :math:`A_i` et :math:`B_i` lignes pour la clé :math:`c_i`. Combien la table finale issue de la fusion des deux tables contiendra-t-elle de lignes ?
pivot (tableau croisé dynamique)¶
Cette opération consiste à créer une seconde table en utilisant utiliser les valeurs d’une colonne comme nom de colonnes.
A |
B |
C |
|---|---|---|
A1 |
B1 |
C1 |
A1 |
B2 |
C2 |
A2 |
B1 |
C3 |
A2 |
B2 |
C4 |
A2 |
B3 |
C5 |
L’opération pivot(A,B,C) donnera :
A |
B1 |
B2 |
B3 |
|---|---|---|---|
A1 |
C1 |
C2 |
|
A2 |
C3 |
C4 |
C5 |
On applique cela aux marathons où on veut avoir les villes comme noms de colonnes et une année par ligne.
[53]:
piv = df.pivot(index="annee", columns="ville", values="temps")
pandas.concat([piv[20:23], piv[40:43], piv.tail(n=3)])
[53]:
| ville | AMSTERDAM | BERLIN | BOSTON | CHICAGO | FUKUOKA | LONDON | NEW YORK | PARIS | STOCKOLM |
|---|---|---|---|---|---|---|---|---|---|
| annee | |||||||||
| 1967 | NaN | NaN | 02:15:45 | NaN | 02:09:37 | NaN | NaN | NaN | NaN |
| 1968 | NaN | NaN | 02:22:17 | NaN | 02:10:48 | NaN | NaN | NaN | NaN |
| 1969 | NaN | NaN | 02:13:49 | NaN | 02:11:13 | NaN | NaN | NaN | NaN |
| 1987 | 02:12:40 | 02:11:11 | 02:11:50 | NaN | 02:08:18 | 02:09:50 | 02:11:01 | 02:11:09 | 02:13:52 |
| 1988 | 02:12:38 | 02:11:45 | 02:08:43 | 02:08:57 | 02:11:04 | 02:10:20 | 02:08:20 | 02:13:53 | 02:14:26 |
| 1989 | 02:13:52 | 02:10:11 | 02:09:06 | 02:11:25 | 02:12:54 | 02:09:03 | 02:08:01 | 02:13:03 | 02:13:34 |
| 2009 | 02:06:18 | 02:06:08 | 02:08:42 | 02:05:41 | 02:05:18 | 02:05:10 | 02:09:15 | 02:05:47 | 02:15:34 |
| 2010 | 02:05:44 | 02:05:08 | 02:05:52 | 02:06:23 | 02:08:24 | 02:05:19 | 02:08:14 | 02:06:41 | 02:12:48 |
| 2011 | NaN | 02:03:38 | 02:03:02 | NaN | NaN | 02:04:40 | NaN | 02:06:29 | 02:14:07 |
Il existe une méthode qui effectue l’opération inverse : Dataframe.stack.
Lambda fonctions¶
Les lambda expressions permettent une syntaxe plus légère (syntactic sugar) pour déclarer une fonction simple. Cela est très utile pour passer une fonction en argument notamment. Par exemple pour trier sur le 2ème element d’un tuple.
[ ]:
pairs = [(1, "one"), (2, "two"), (3, "three"), (4, "four")]
pairs.sort(key=lambda pair: pair[1])
print(pairs)
[(4, 'four'), (1, 'one'), (3, 'three'), (2, 'two')]
On peut réécrire le groupby aggrégé par max_entier en utilisant une fonction lambda
[55]:
def max_entier(x):
return int(max(x))
nb = df[["annee", "secondes"]].groupby("annee").agg(max_entier).reset_index()
nb.tail(n=3)
# same as:
nb = df[["annee", "secondes"]].groupby("annee").agg(lambda x: int(max(x))).reset_index()
nb.tail(n=3)
[55]:
| annee | secondes | |
|---|---|---|
| 62 | 2009 | 8134 |
| 63 | 2010 | 7968 |
| 64 | 2011 | 8047 |
Exercice 2 : lambda fonction¶
Ecrire une lambda fonction qui prend deux paramètres et qui est équivalente à la fonction suivante :
[ ]:
def delta(x, y):
return max(x, y) - min(x, y)
On utilise beaucoup les lambda fonctions lorsqu’une fonction prend une fonction en argument :
[ ]:
def riemann(a, b, f, n):
return sum(f(a + (b - a) * i / n) for i in range(0, n)) / n
riemann(0, 1, lambda x: x**2, 1000)
0.3328335
Ensuite, il faut utiliser une lambda fonction et la fonction apply pour tirer un échantillon aléatoire
Exercice 3 : moyennes par groupes¶
Toujours avec le même jeu de données (marathon.txt), on veut ajouter une ligne à la fin du tableau croisé dynamique contenant la moyenne en secondes des temps des marathons pour chaque ville.
[ ]:
Avec ou sans index¶
Une façon naïve de faire une jointure entre deux tables de taille et
et de regarder toutes les
combinaisons possibles. La taille de la table résultante dépend du type de jointure (
inner, outer) et de l’unicité des clés utilisées pour la jointure. Si les clés sont uniques, la table finale aura au plus lignes (une par clé).
Dans la plupart des cas, opérations est beaucoup trop long. On peut faire plus rapide en triant chacune des tables d’abord et en les fusionnant :
. Si
, il est évident que cette façon de faire est plus rapide. C’est une des choses que fait pandas (présentation) (voir aussi klib).
On peut trier une table selon une clé ou encore utiliser une table de hachage), il est alors très rapide de retrouver la ligne ou les lignes qui partagent cette clé. On dit que la table est indexée selon cette clé. Indexer selon une ou plusieurs colonnes une table accélère toute opération s’appuyant sur ces colonnes comme la recherche d’un élément.
On veut comparer le temps nécessaire pour une recherche. Pour cela on utilise la %magic function %timeit (ou %%timeit si on veut l”appliquer à la cellule) de Jupyter.
[58]:
import pandas, random
big_df = pandas.DataFrame(
{
"cle1": random.randint(1, 100),
"cle2": random.randint(1, 100),
"autre": random.randint(1, 10),
}
for i in range(0, 100000)
)
big_df.shape
[58]:
(100000, 3)
[59]:
%timeit big_df[(big_df.cle1 == 1) & (big_df.cle2 == 1)]
398 μs ± 10.1 μs per loop (mean ± std. dev. of 7 runs, 1,000 loops each)
Et la version indexée :
[60]:
big_dfi = big_df.set_index(["cle1", "cle2"])
big_dfi = big_dfi.sort_index() # Il ne faut oublier de trier.
%timeit big_dfi.loc[(1,1), :]
139 μs ± 4.99 μs per loop (mean ± std. dev. of 7 runs, 10,000 loops each)
[61]:
big_dfi.head()
[61]:
| autre | ||
|---|---|---|
| cle1 | cle2 | |
| 1 | 1 | 8 |
| 1 | 5 | |
| 1 | 10 | |
| 1 | 6 | |
| 1 | 7 |
Plus la table est grande, plus le gain est important.
Dates¶
Les dates sont souvent compliquées à gérer car on n’utilise pas le mêmes format dans tous les pays. Pour faire simple, je recommande deux options :
Soit convertir les dates/heures au format chaînes de caractères
AAAA-MM-JJ hh:mm:ss:msqui permet de trier les dates par ordre croissant.Soit convertir les dates/heures au format datetime (date) ou timedelta (durée), voir format de date/heure).
Par exemple, voici le code qui a permis de générer la colonne seconde de la table marathon :
[62]:
from datetime import datetime, time
df = df_marathon
df = df[["ville", "annee", "temps"]] # on enlève la colonne secondes pour la recréer
df["secondes"] = df.apply(
lambda r: (
datetime.strptime(r.temps, "%H:%M:%S") - datetime(1900, 1, 1)
).total_seconds(),
axis=1,
)
df.head()
[62]:
| ville | annee | temps | secondes | |
|---|---|---|---|---|
| 0 | PARIS | 2011 | 02:06:29 | 7589.0 |
| 1 | PARIS | 2010 | 02:06:41 | 7601.0 |
| 2 | PARIS | 2009 | 02:05:47 | 7547.0 |
| 3 | PARIS | 2008 | 02:06:40 | 7600.0 |
| 4 | PARIS | 2007 | 02:07:17 | 7637.0 |
Plot(s)¶
Récupération des données¶
On récupère les données disponibles sur le site de l’INSEE : Naissance, décès, mariages 2012. Il s’agit de récupérer la liste des mariages de l’année 2012. On souhaite représenter le graphe du nombre de mariages en fonction de l’écart entre les mariés.
[63]:
import urllib.request
import zipfile
import http.client
def download_and_save(name, root_url):
try:
response = urllib.request.urlopen(root_url + name)
except (TimeoutError, urllib.request.URLError, http.client.BadStatusLine):
# back up plan
root_url = (
"https://github.com/sdpython/teachdata/raw/main/naissance_deces_mariage/"
)
response = urllib.request.urlopen(root_url + name)
with open(name, "wb") as outfile:
outfile.write(response.read())
def unzip(name):
with zipfile.ZipFile(name, "r") as z:
z.extractall(".")
filenames = [
"etatcivil2012_mar2012_dbase.zip",
"etatcivil2012_nais2012_dbase.zip",
"etatcivil2012_dec2012_dbase.zip",
]
root_url = "https://www.insee.fr/fr/statistiques/fichier/2407910/"
for filename in filenames:
download_and_save(filename, root_url)
unzip(filename)
print("Download of {}: DONE!".format(filename))
Download of etatcivil2012_mar2012_dbase.zip: DONE!
Download of etatcivil2012_nais2012_dbase.zip: DONE!
Download of etatcivil2012_dec2012_dbase.zip: DONE!
L’exemple suivant pourrait ne pas marcher si le module dbfread n’est pas installé. Si tel est le cas, le programme utilisera une version des données après utilisation de ce module.
[64]:
import pandas
from dbfread import DBF
def dBase2df(dbase_filename):
table = DBF(dbase_filename, load=True, encoding="cp437")
return pandas.DataFrame(table.records)
df = df_civil = dBase2df("mar2012.dbf")
# df.to_csv("mar2012.txt", sep="\t", encoding="utf8", index=False)
df.shape, df.columns
[64]:
((246123, 16),
Index(['ANAISH', 'DEPNAISH', 'INDNATH', 'ETAMATH', 'ANAISF', 'DEPNAISF',
'INDNATF', 'ETAMATF', 'AMAR', 'MMAR', 'JSEMAINE', 'DEPMAR', 'DEPDOM',
'TUDOM', 'TUCOM', 'NBENFCOM'],
dtype='str'))
L”encoding est une façon de représenter les caractères spéciaux (comme les caractères accentuées). L’encoding le plus répandu est utf-8. Sans la mention encoding="cp437", la fonction qui lit le fichier fait des erreurs lors de la lecture car elle ne sait pas comment interpréter certains caractères spéciaux. On récupère de la même manière la signification des variables :
[65]:
vardf = dBase2df("varlist_mariages.dbf")
print(vardf.shape, vardf.columns)
vardf
(16, 4) Index(['VARIABLE', 'LIBELLE', 'TYPE', 'LONGUEUR'], dtype='str')
[65]:
| VARIABLE | LIBELLE | TYPE | LONGUEUR | |
|---|---|---|---|---|
| 0 | AMAR | Année du mariage | CHAR | 4 |
| 1 | ANAISF | Année de naissance de l'épouse | CHAR | 4 |
| 2 | ANAISH | Année de naissance de l'époux | CHAR | 4 |
| 3 | DEPDOM | Département de domicile après le mariage | CHAR | 3 |
| 4 | DEPMAR | Département de mariage | CHAR | 3 |
| 5 | DEPNAISF | Département de naissance de l'épouse | CHAR | 3 |
| 6 | DEPNAISH | Département de naissance de l'époux | CHAR | 3 |
| 7 | ETAMATF | État matrimonial antérieur de l'épouse | CHAR | 1 |
| 8 | ETAMATH | État matrimonial antérieur de l'époux | CHAR | 1 |
| 9 | INDNATF | Indicateur de nationalité de l'épouse | CHAR | 1 |
| 10 | INDNATH | Indicateur de nationalité de l'époux | CHAR | 1 |
| 11 | JSEMAINE | Jour du mariage dans la semaine | CHAR | 1 |
| 12 | MMAR | Mois du mariage | CHAR | 2 |
| 13 | NBENFCOM | Enfants en commun avant le mariage | CHAR | 1 |
| 14 | TUCOM | Tranche de commune du lieu de domicile des époux | CHAR | 1 |
| 15 | TUDOM | Tranche d'unité urbaine du lieu de domicile de... | CHAR | 1 |
Exercice 4 : nuage de points¶
On veut tracer un nuage de points avec en abscisse l’âge du mari, en ordonnée, l’âge de la femme. Il faudra peut-être jeter un coup d’oeil sur la documentation de la méthode plot. Etant donné le nombre d’observations, ce graphe risque d’être moins lisible qu’une heatmap.
[66]:
# df.plot(...)
Exercice 5 : graphe d’une distribution avec pandas¶
En ajoutant une colonne et en utilisant l’opération group by, on veut obtenir la distribution du nombre de mariages en fonction de l’écart entre les mariés. Au besoin, on changera le type d’une colone ou deux. Le module pandas propose un panel de graphiques standard faciles à obtenir. On souhaite représenter la distribution sous forme d’histogramme. A vous de choisir le meilleure graphique depuis la page
Visualization.
[67]:
# df["colonne"] = df.apply(lambda r: int(r["colonne"]), axis=1) # pour changer de type
# df["difference"] = ...
matplotlib¶
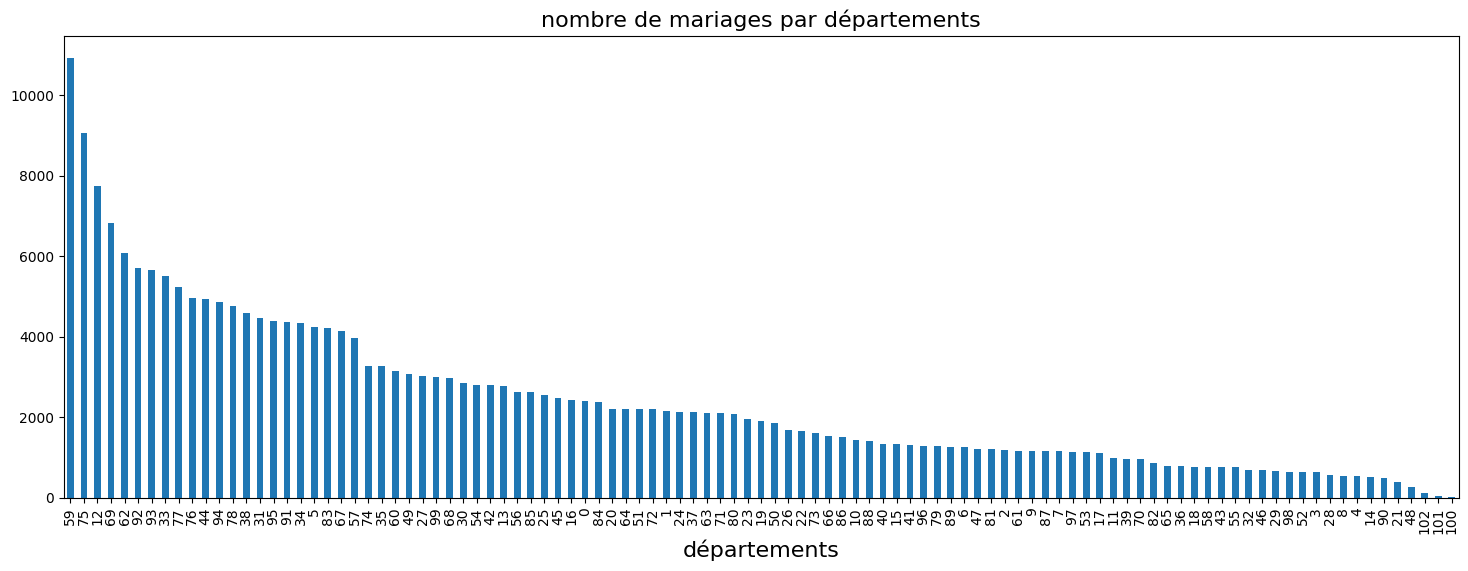
matplotlib est le module qu’utilise pandas. Ainsi, la méthode plot retourne un objet de type Axes qu’on peut modifier par la suite via les méthodes suivantes. On peut ajouter un titre avec set_title ou ajouter une grille avec grid. On peut également superposer deux courbes sur le même graphique, ou changer de taille de caractères. Le code suivant trace le nombre de mariages par département.
[68]:
df["nb"] = 1
dep = (
df[["DEPMAR", "nb"]]
.groupby("DEPMAR", as_index=False)
.sum()
.sort_values("nb", ascending=False)
)
ax = dep.plot(kind="bar", figsize=(18, 6))
ax.set_xlabel("départements", fontsize=16)
ax.set_title("nombre de mariages par départements", fontsize=16)
ax.legend().set_visible(False) # on supprime la légende
Quand on ne sait pas, le plus simple est d’utiliser un moteur de recherche avec un requête du type : matplotlib + requête. Pour créer un graphique, le plus courant est de choisir le graphique le plus ressemblant d’une gallerie de graphes puis de l’adapter à vos données.
Exercice 6 : distribution des mariages par jour¶
On veut obtenir un graphe qui contient l’histogramme de la distribution du nombre de mariages par jour de la semaine et d’ajouter une seconde courbe correspond avec un second axe à la répartition cumulée.
[ ]:
Annexes¶
Créer un fichier Excel avec plusieurs feuilles¶
La page Allow ExcelWriter() to add sheets to existing workbook donne plusieurs exemples d’écriture. On diminue la taille du document Excel à écrire.
[69]:
df1000 = df[:1000]
[71]:
import pandas
writer = pandas.ExcelWriter("ton_example100.xlsx")
df1000.to_excel(writer, sheet_name="Data 0")
df1000.to_excel(writer, sheet_name="Data 1")
writer.close()
Eléments de réponses pour les exercices¶
Exercice 1 : créer un fichier Excel¶
On souhaite récupérer les données donnees_enquete_2003_television.txt.
POIDSLOG: Pondération individuelle relativePOIDSF: Variable de pondération individuellecLT1FREQ: Nombre d’heures en moyenne passées à regarder la télévisioncLT2FREQ: Unité de temps utilisée pour compter le nombre d’heures passées à regarder la télévision, cette unité est représentée par les quatre valeurs suivantes0 : non concerné
1 : jour
2 : semaine
3 : mois
Ensuite, on veut :
Supprimer les colonnes vides
Obtenir les valeurs distinctes pour la colonne
cLT2FREQModifier la matrice pour enlever les lignes pour lesquelles l’unité de temps (cLT2FREQ) n’est pas renseignée ou égale à zéro.
Sauver le résultat au format Excel.
Vous aurez peut-être besoin des fonctions suivantes :
[72]:
df = pandas.read_csv(
"https://github.com/sdpython/teachpyx/raw/main/_data/donnees_enquete_2003_television.txt",
sep="\t",
)
df.head()
[72]:
| POIDLOG | POIDSF | cLT1FREQ | cLT2FREQ | Unnamed: 4 | Unnamed: 5 | Unnamed: 6 | Unnamed: 7 | Unnamed: 8 | Unnamed: 9 | ... | Unnamed: 22 | Unnamed: 23 | Unnamed: 24 | Unnamed: 25 | Unnamed: 26 | Unnamed: 27 | Unnamed: 28 | Unnamed: 29 | Unnamed: 30 | Unnamed: 31 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0.889422 | 4766.865201 | 2 | 1.0 | NaN | NaN | NaN | NaN | NaN | NaN | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN |
| 1 | 2.310209 | 12381.589746 | 30 | 1.0 | NaN | NaN | NaN | NaN | NaN | NaN | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN |
| 2 | 2.740070 | 14685.431344 | 6 | 2.0 | NaN | NaN | NaN | NaN | NaN | NaN | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN |
| 3 | 1.775545 | 9516.049939 | 1 | 1.0 | NaN | NaN | NaN | NaN | NaN | NaN | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN |
| 4 | 0.732512 | 3925.907588 | 3 | 1.0 | NaN | NaN | NaN | NaN | NaN | NaN | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN |
5 rows × 32 columns
On enlève les colonnes vides :
[73]:
df = df[[c for c in df.columns if "Unnamed" not in c]]
df.head()
[73]:
| POIDLOG | POIDSF | cLT1FREQ | cLT2FREQ | |
|---|---|---|---|---|
| 0 | 0.889422 | 4766.865201 | 2 | 1.0 |
| 1 | 2.310209 | 12381.589746 | 30 | 1.0 |
| 2 | 2.740070 | 14685.431344 | 6 | 2.0 |
| 3 | 1.775545 | 9516.049939 | 1 | 1.0 |
| 4 | 0.732512 | 3925.907588 | 3 | 1.0 |
[74]:
notnull = df[~df.cLT2FREQ.isnull()] # équivalent ) df [ df.cLT2FREQ.notnull() ]
print(len(df), len(notnull))
notnull.tail()
8403 7386
[74]:
| POIDLOG | POIDSF | cLT1FREQ | cLT2FREQ | |
|---|---|---|---|---|
| 8397 | 0.502091 | 2690.961176 | 3 | 1.0 |
| 8398 | 0.306852 | 1644.574141 | 6 | 1.0 |
| 8399 | 2.501181 | 13405.104689 | 6 | 1.0 |
| 8400 | 1.382758 | 7410.905653 | 1 | 1.0 |
| 8401 | 0.343340 | 1840.132652 | 3 | 1.0 |
[75]:
notnull.to_excel("data.xlsx") # question 4
Questions
Que changerait l’ajout du paramètre
how='outer'dans ce cas ?On cherche à joindre deux tables A,B qui ont chacune trois clés distinctes :
. Il y a respectivement dans chaque table
et
lignes pour la clé
. Combien la table finale issue de la fusion des deux tables contiendra-t-elle de lignes ?
L’ajout du paramètres how='outer' ne changerait rien dans ce cas car les deux tables fusionnées contiennent exactement les mêmes clés.
Le nombre de lignes obtenus serait . Il y a trois clés, chaque ligne de la table A doit être associée à toutes les lignes de la table B partageant la même clé.
Exercice 2 : lambda function¶
Ecrire une lambda function qui prend deux paramètres et qui est équivalente à la fonction suivante
[76]:
def delta(x, y):
return max(x, y) - min(x, y)
[77]:
delta = lambda x, y: max(x, y) - min(x, y)
[78]:
delta(4, 5)
[78]:
1
[79]:
import random
df["select"] = df.apply(lambda row: random.randint(1, 10), axis=1)
echantillon = df[df["select"] == 1]
echantillon.shape, df.shape
[79]:
((892, 5), (8403, 5))
Exercice 3 : moyennes par groupes¶
Toujours avec le même jeu de données, on veut ajouter une ligne à la fin du tableau croisé dynamique contenant la moyenne en secondes des temps des marathons pour chaque ville.
La solution requiert trois étapes.
Pour avoir la moyenne par villes, il faut grouper les lignes associées à la même villes.
Ensuite, il faut introduire ces moyennes dans la table initiale : on fusionne.
On effectue le même pivot que dans l’énoncé.
[80]:
# étape 1
# par défaut, la méthode groupby utilise la clé de group comme index
# pour ne pas le faire, il faut préciser as_index = False
df = df_marathon
gr = df[["ville", "secondes"]].groupby("ville", as_index=False).mean()
gr.head()
[80]:
| ville | secondes | |
|---|---|---|
| 0 | AMSTERDAM | 7883.371429 |
| 1 | BERLIN | 7922.315789 |
| 2 | BOSTON | 7891.061224 |
| 3 | CHICAGO | 7815.909091 |
| 4 | FUKUOKA | 8075.187500 |
[81]:
# étape 2 - on ajoute une colonne
tout = df.merge(gr, on="ville")
tout.head()
[81]:
| ville | annee | temps | secondes_x | secondes_y | |
|---|---|---|---|---|---|
| 0 | PARIS | 2011 | 02:06:29 | 7589 | 7937.028571 |
| 1 | PARIS | 2010 | 02:06:41 | 7601 | 7937.028571 |
| 2 | PARIS | 2009 | 02:05:47 | 7547 | 7937.028571 |
| 3 | PARIS | 2008 | 02:06:40 | 7600 | 7937.028571 |
| 4 | PARIS | 2007 | 02:07:17 | 7637 | 7937.028571 |
[82]:
# étape 3
piv = tout.pivot(index="annee", columns="ville", values="secondes_x")
piv.tail()
[82]:
| ville | AMSTERDAM | BERLIN | BOSTON | CHICAGO | FUKUOKA | LONDON | NEW YORK | PARIS | STOCKOLM |
|---|---|---|---|---|---|---|---|---|---|
| annee | |||||||||
| 2007 | 7589.0 | 7466.0 | 8053.0 | 7871.0 | 7599.0 | 7661.0 | 7744.0 | 7637.0 | 8456.0 |
| 2008 | 7672.0 | 7439.0 | 7665.0 | 7585.0 | 7570.0 | 7515.0 | 7723.0 | 7600.0 | 8163.0 |
| 2009 | 7578.0 | 7568.0 | 7722.0 | 7541.0 | 7518.0 | 7510.0 | 7755.0 | 7547.0 | 8134.0 |
| 2010 | 7544.0 | 7508.0 | 7552.0 | 7583.0 | 7704.0 | 7519.0 | 7694.0 | 7601.0 | 7968.0 |
| 2011 | NaN | 7418.0 | 7382.0 | NaN | NaN | 7480.0 | NaN | 7589.0 | 8047.0 |
A partir de là, on ne voit pas trop comment s’en sortir. Voici ce que je propose :
On effectue un pivot sur la petite matrice des moyennes.
On ajoute ce second pivot avec le premier (celui de l’énoncé).
[83]:
gr["annee"] = "moyenne"
pivmean = gr.pivot(index="annee", values="ville", columns="secondes")
pivmean
[83]:
| secondes | 7695.161290 | 7815.909091 | 7883.371429 | 7891.061224 | 7922.315789 | 7928.560976 | 7937.028571 | 8075.187500 | 8133.393939 |
|---|---|---|---|---|---|---|---|---|---|
| annee | |||||||||
| moyenne | LONDON | CHICAGO | AMSTERDAM | BOSTON | BERLIN | NEW YORK | PARIS | FUKUOKA | STOCKOLM |
[84]:
piv = df.pivot(index="annee", columns="ville", values="secondes")
pandas.concat([piv, pivmean]).tail()
[84]:
| AMSTERDAM | BERLIN | BOSTON | CHICAGO | FUKUOKA | LONDON | NEW YORK | PARIS | STOCKOLM | 7695.16129 | 7815.909091 | 7883.371429 | 7891.061224 | 7922.315789 | 7928.560976 | 7937.028571 | 8075.1875 | 8133.393939 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| annee | ||||||||||||||||||
| 2008 | 7672.0 | 7439.0 | 7665.0 | 7585.0 | 7570.0 | 7515.0 | 7723.0 | 7600.0 | 8163.0 | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN |
| 2009 | 7578.0 | 7568.0 | 7722.0 | 7541.0 | 7518.0 | 7510.0 | 7755.0 | 7547.0 | 8134.0 | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN |
| 2010 | 7544.0 | 7508.0 | 7552.0 | 7583.0 | 7704.0 | 7519.0 | 7694.0 | 7601.0 | 7968.0 | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN |
| 2011 | NaN | 7418.0 | 7382.0 | NaN | NaN | 7480.0 | NaN | 7589.0 | 8047.0 | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN |
| moyenne | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | LONDON | CHICAGO | AMSTERDAM | BOSTON | BERLIN | NEW YORK | PARIS | FUKUOKA | STOCKOLM |
En résumé, cela donne (j’ajoute aussi le nombre de marathons courus) :
[85]:
piv = df.pivot(index="annee", columns="ville", values="secondes")
gr = df[["ville", "secondes"]].groupby("ville", as_index=False).mean()
gr["annee"] = "moyenne"
pivmean = gr.pivot(index="annee", columns="ville", values="secondes")
pandas.concat([piv, pivmean]).tail()
[85]:
| ville | AMSTERDAM | BERLIN | BOSTON | CHICAGO | FUKUOKA | LONDON | NEW YORK | PARIS | STOCKOLM |
|---|---|---|---|---|---|---|---|---|---|
| annee | |||||||||
| 2008 | 7672.000000 | 7439.000000 | 7665.000000 | 7585.000000 | 7570.0000 | 7515.00000 | 7723.000000 | 7600.000000 | 8163.000000 |
| 2009 | 7578.000000 | 7568.000000 | 7722.000000 | 7541.000000 | 7518.0000 | 7510.00000 | 7755.000000 | 7547.000000 | 8134.000000 |
| 2010 | 7544.000000 | 7508.000000 | 7552.000000 | 7583.000000 | 7704.0000 | 7519.00000 | 7694.000000 | 7601.000000 | 7968.000000 |
| 2011 | NaN | 7418.000000 | 7382.000000 | NaN | NaN | 7480.00000 | NaN | 7589.000000 | 8047.000000 |
| moyenne | 7883.371429 | 7922.315789 | 7891.061224 | 7815.909091 | 8075.1875 | 7695.16129 | 7928.560976 | 7937.028571 | 8133.393939 |
Exercice 4 : écart entre les mariés¶
En ajoutant une colonne et en utilisant l’opération group by, on veut obtenir la distribution du nombre de mariages en fonction de l’écart entre les mariés. Au besoin, on changera le type d’une colone ou deux.
On veut tracer un nuage de points avec en abscisse l’âge du mari, en ordonnée, l’âge de la femme. Il faudra peut-être jeter un coup d’oeil sur la documentation de la méthode plot.
[86]:
df = df_civil # on récupère le dataframe déjà construit
print(df.shape, df.columns)
df.head()
(246123, 17) Index(['ANAISH', 'DEPNAISH', 'INDNATH', 'ETAMATH', 'ANAISF', 'DEPNAISF',
'INDNATF', 'ETAMATF', 'AMAR', 'MMAR', 'JSEMAINE', 'DEPMAR', 'DEPDOM',
'TUDOM', 'TUCOM', 'NBENFCOM', 'nb'],
dtype='str')
[86]:
| ANAISH | DEPNAISH | INDNATH | ETAMATH | ANAISF | DEPNAISF | INDNATF | ETAMATF | AMAR | MMAR | JSEMAINE | DEPMAR | DEPDOM | TUDOM | TUCOM | NBENFCOM | nb | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1982 | 75 | 1 | 1 | 1984 | 99 | 2 | 1 | 2012 | 01 | 1 | 29 | 99 | 9 | N | 1 | |
| 1 | 1956 | 69 | 2 | 4 | 1969 | 99 | 2 | 4 | 2012 | 01 | 3 | 75 | 99 | 9 | N | 1 | |
| 2 | 1982 | 99 | 2 | 1 | 1992 | 99 | 1 | 1 | 2012 | 01 | 5 | 34 | 99 | 9 | N | 1 | |
| 3 | 1985 | 99 | 2 | 1 | 1987 | 84 | 1 | 1 | 2012 | 01 | 4 | 13 | 99 | 9 | N | 1 | |
| 4 | 1968 | 99 | 2 | 1 | 1963 | 99 | 2 | 1 | 2012 | 01 | 6 | 26 | 99 | 9 | N | 1 |
[87]:
df["ageH"] = df.apply(lambda r: 2014 - int(r["ANAISH"]), axis=1)
df["ageF"] = df.apply(lambda r: 2014 - int(r["ANAISF"]), axis=1)
df.head()
[87]:
| ANAISH | DEPNAISH | INDNATH | ETAMATH | ANAISF | DEPNAISF | INDNATF | ETAMATF | AMAR | MMAR | JSEMAINE | DEPMAR | DEPDOM | TUDOM | TUCOM | NBENFCOM | nb | ageH | ageF | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1982 | 75 | 1 | 1 | 1984 | 99 | 2 | 1 | 2012 | 01 | 1 | 29 | 99 | 9 | N | 1 | 32 | 30 | |
| 1 | 1956 | 69 | 2 | 4 | 1969 | 99 | 2 | 4 | 2012 | 01 | 3 | 75 | 99 | 9 | N | 1 | 58 | 45 | |
| 2 | 1982 | 99 | 2 | 1 | 1992 | 99 | 1 | 1 | 2012 | 01 | 5 | 34 | 99 | 9 | N | 1 | 32 | 22 | |
| 3 | 1985 | 99 | 2 | 1 | 1987 | 84 | 1 | 1 | 2012 | 01 | 4 | 13 | 99 | 9 | N | 1 | 29 | 27 | |
| 4 | 1968 | 99 | 2 | 1 | 1963 | 99 | 2 | 1 | 2012 | 01 | 6 | 26 | 99 | 9 | N | 1 | 46 | 51 |
[88]:
df.plot(x="ageH", y="ageF", kind="scatter");
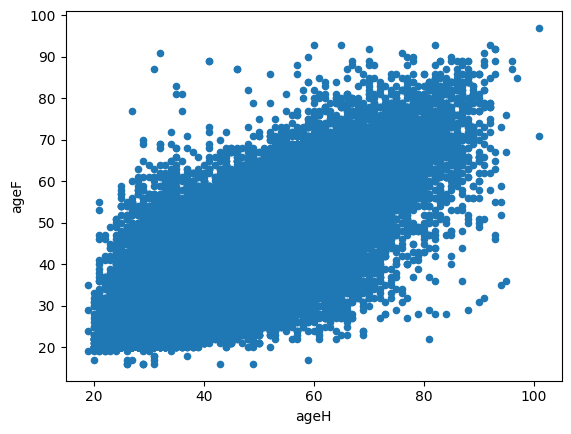
[89]:
df.plot(x="ageH", y="ageF", kind="hexbin");
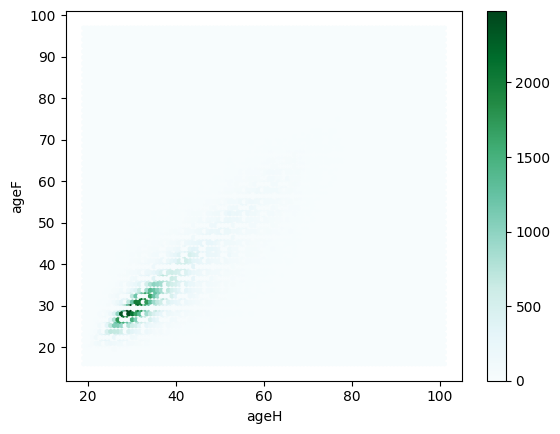
Exercice 5 : graphe de la distribution avec pandas¶
Le module pandas propose un panel de graphiques standard faciles à obtenir. On souhaite représenter la distribution sous forme d’histogramme. A vous de choisir le meilleure graphique depuis la page Visualization.
[90]:
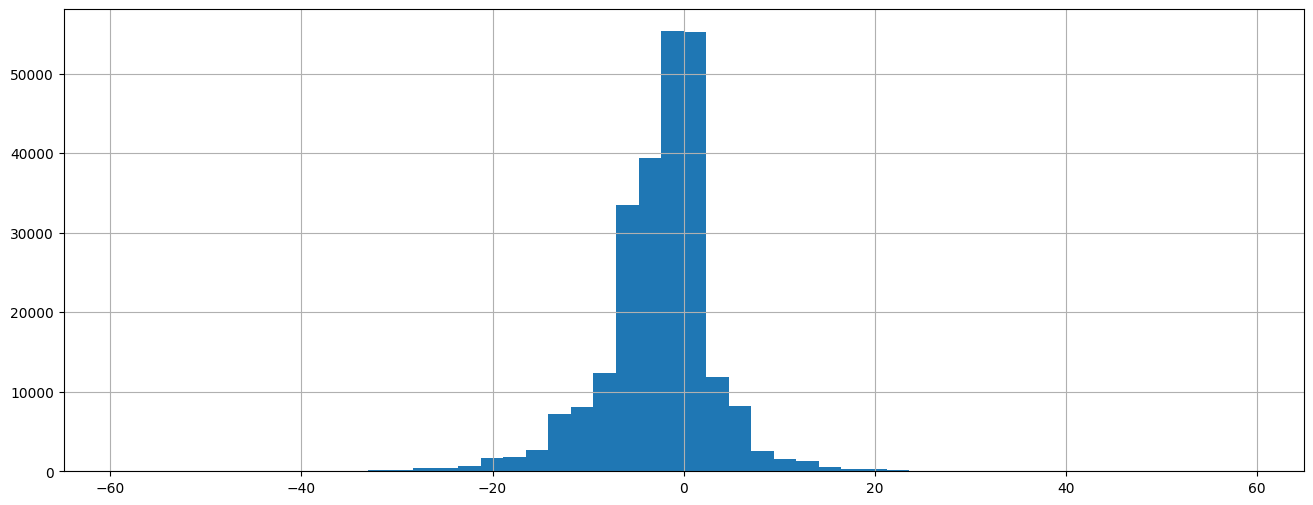
df["ANAISH"] = df.apply(lambda r: int(r["ANAISH"]), axis=1)
df["ANAISF"] = df.apply(lambda r: int(r["ANAISF"]), axis=1)
df["differenceHF"] = df.ANAISH - df.ANAISF
df["nb"] = 1
dist = df[["nb", "differenceHF"]].groupby("differenceHF", as_index=False).count()
df["differenceHF"].hist(figsize=(16, 6), bins=50);
Exercice 6 : distribution des mariages par jour¶
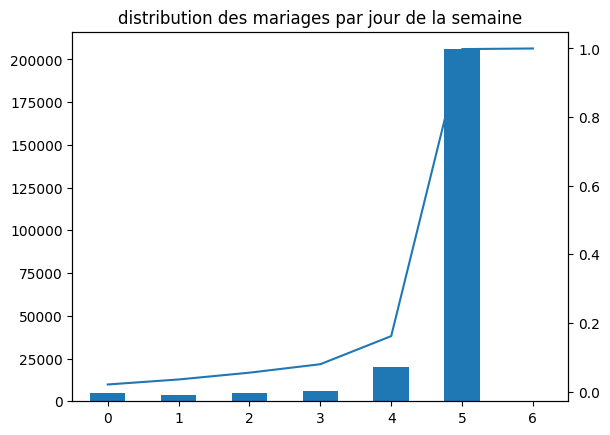
On veut obtenir un graphe qui contient l’histogramme de la distribution du nombre de mariages par jour de la semaine et d’ajouter une seconde courbe correspond avec un second axe à la répartition cumulée.
[91]:
df["nb"] = 1
dissem = df[["JSEMAINE", "nb"]].groupby("JSEMAINE", as_index=False).sum()
total = dissem["nb"].sum()
repsem = dissem.cumsum()
repsem["nb"] /= total
ax = dissem["nb"].plot(kind="bar")
repsem["nb"].plot(ax=ax, secondary_y=True)
ax.set_title("distribution des mariages par jour de la semaine");